Premio Nobel de Química 2020: Herramientas para la edición genómica
F. Alejandro Sánchez Flores
Miembro de la Academia de Ciencias de Morelos
Los premios Nobel fueron el último deseo antes de morir, del sueco Alfred Nobel quien nació en Estocolmo Suecia en 1833. Fue un científico, inventor, escritor, hombre de negocios y fabricante de armas, quien además de haber inventado la dinamita y haber registrado 355 patentes, también estaba muy interesado en las causas sociales y la paz. En noviembre de 1895, un año antes fallecer, firmó su testamento donde estipula que con su fortuna se creara un fondo con el que se premiaría a personas cuyo trabajo haya contribuido al beneficio de la humanidad. En 1900 los albaceas de la fortuna de Alfred formaron la Fundación Nobel cuya misión es gestionar las finanzas de tal forma que permita asegurar los premios a largo plazo. Fue hasta 1901 que se entregan los primeros galardones. El 7 de octubre del 2020, la Real Academia de las Ciencias de Suecia anunció que el premio Nobel en el área de Química, fue para las investigadoras Emmanuelle Charpentier de la Max Planck Unit for the Science of Pathogens ubicada en Berlín, Alemania y Jennifer A. Doudna de la Universidad de California en Berkeley, Estados Unidos. Su trabajo relacionado con el desarrollo de un método para la edición genómica, ha revolucionado el mundo de la ciencia en relativamente poco tiempo. En esta entrega, hacemos una breve reseña del trabajo de estas investigadoras y como esto a contribuido al desarrollo de la ciencia y al cambio de muchos paradigmas.
El “cut and paste” para el ADN
Un genoma es el compendio de la información genética de cualquier organismo y esta información se encuentra en las moléculas de ADN que contiene cada célula de dicho organismo. Además, si recordamos que esta molécula está formada por cuatro bloques básicos que pueden ser representados por las letras A, T, C y C, es posible representar como un largo texto la información genética. Ahora, imaginemos que es posible tener las mismas herramientas que tenemos para un editor de textos en nuestra computadora. Una de las funciones mas cómodas es la de copiar y pegar (cut and paste en inglés) ya que es de gran utilidad para mover o reacomodar información en un escrito, sin tener que borrar o volver a escribir. De manera análoga, el trabajo de Charpentier y Doudna (Figura 1) ha proporcionado una herramienta molecular que permite modificar la información genética de cualquier organismo. Si bien las aplicaciones son muchas, empezaremos por explicar cual es el mecanismo de estas “tijeras” moleculares para ADN, llamada CRISPR-Cas9.

Figura 1. Ilustración de Emmanuelle Charpentier (izquierda) y Jennifer Doudna (derecha), ganadoras del premio Nobel de Química en el 2020. Tomada de: https://www.nationalgeographic.com.es/ciencia/premio-nobel-quimica-2020-para-inventoras-tijeras-geneticas_15961
Repeticiones en la información genética de las bacterias
En 1987, un grupo de investigadores japoneses descubrieron en el genoma de la bacteria Escherichia coli, unas secuencias que se repetían de manera regular y que estaban separadas unas de otras a la misma distancia. De manera más precisa, encontraron secuencias de 29 letras (pares de bases) que se repetían varias veces con una misma orientación. Dentro de esta repetición, observaron una región palíndroma que correspondía a la duplicación de una secuencia de 7 letras orientada en sentido inverso una respecto a la otra. Un palíndromo es una palabra que podemos leer de la misma manera de atrás hacia adelante (por ejemplo, RECONOCER). Además, las repeticiones presentaban la singularidad de que la distancia entre ellas era de 32 letras o en algunos casos hasta 33 letras. Más tarde, en 1993 y de forma independiente, el científico Francisco J. M. Mojica de la Universidad de Alicante, descubre en la arqueobacteria Haloferax mediterranei, el mismo tipo de arreglo. En el año 2000, Francisco J. M. Mojica y colaboradores detectaron un gran número de estas secuencias repetidas en otras bacterias, arqueas e incluso mitocondrias (organelos de células eucariontes). A este arreglo en el genoma le llamaron CRISPR que son las siglas en inglés de clustered regularly interspaced short palindromic repeats (en español, repeticiones palindrómicas cortas agrupadas y regularmente interespaciadas).
CRISPR y el sistema inmune en bacterias
Nuevamente fue el grupo de Francisco J. Mojica quienes se dieron cuenta de la relación que existe entre las secuencias CRISPR y los espaciadores asociados, con lo que podría ser considerado un análogo del sistema inmune en bacterias. Sus observaciones fueron clave para la caracterización del sistema CRISPR y las proteínas Cas. Esta familia de proteínas tiene una actividad de endonucleasa, lo cual les permite hacer cortes en alguna parte de una cadena de ADN. Sin embargo, la diferencia con respecto a otras proteínas con la misma actividad, es que estas enzimas pueden usar una “guía” para realizar dichos cortes sobre las cadenas de ADN.
Entonces, las bacterias pueden capturar ADN del exterior el cual normalmente degradan y aprovechar las moléculas que forman la cadena. Pero también las bacterias son víctimas de virus o plásmidos que las infectan para poder replicarse. En particular, a los virus que infectan bacterias se les llama bacteriófagos. Por lo tanto, las bacterias requieren de un sistema que les permita reconocer y diferenciar el ADN extraño del propio para así poder defenderse de estas infecciones. Una manera de hacerlo es capturar el ADN invasor e integrarlo en las regiones CRISPR en forma de un espaciador. Esta sería la primera etapa de su respuesta inmune. La siguiente etapa es transcribir esa información para crear moléculas de ARN y procesarlas para tener pequeños fragmentos muestra de la secuencia del ADN invasor. El tener esas copias de ADN invasor integrado a su propia información genética, funciona de manera similar a tener una “memoria inmune” y con los fragmentos de ARN producidos, otras proteínas Cas pueden usarlos como guía para degradar ADN o ARN externo, ya que quedaría reconocido gracias a las “guías” de ARN, como material genético invasor (Figura 2). De esta manera, las bacterias se protegen de las infecciones por bacteriófagos y entre más secuencias externas guarden en sus arreglos CRISPR, mejor protegidas están contra los ataques de virus. A este proceso se le conoce como interferencia por ARN.
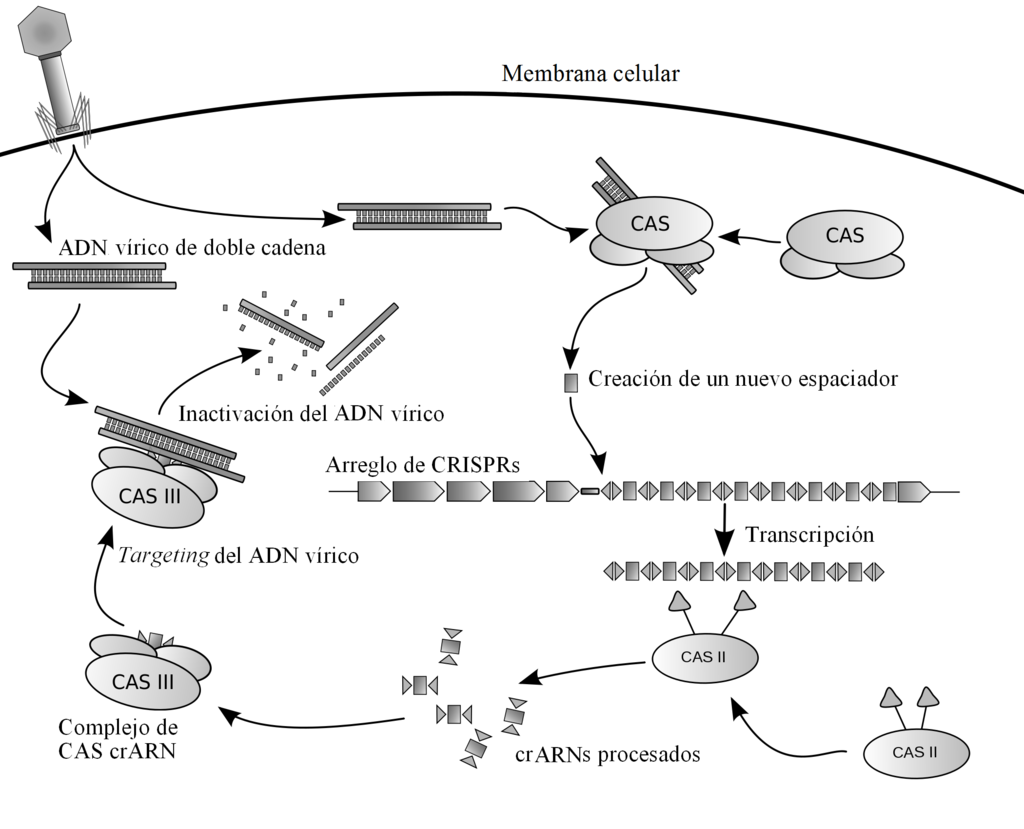
Figura 2. Sistema CRISPR-Cas en bacterias. Si un bacteriófago inyecta su material genético, las proteínas Cas son capaces de integrarlos como un espaciador en sus arreglos CRISPR. Estas secuencias son transcritas y utilizadas como “guías” para que otras proteínas Cas encuentren y destruyan ADN de bacteriófagos que traten de invadir a la bacteria, para así degradarlo y que no cause daño. Tomada de: https://es.wikipedia.org/wiki/CRISPR#/media/Archivo:CRISPR_espa%C3%B1ol.png
El sistema CRISPR-Cas9 y el comienzo de la edición genética
En particular, el investigador Moineau y sus colegas caracterizaron una combinación de arreglos de ADN con una proteína llamada Cas9 y llamaron a este sistema CRISPR-Cas9. Ellos demostraron que la proteína crea cortes en posiciones precisas de las cadenas dobles del ADN, cuando es reconocida por la secuencia guía que se obtiene de los espaciadores en las regiones CRISPR. Este corte es de 3 nucleótidos hacia arriba de las regiones repetitivas del arreglo. Esta es una característica distintiva de los sistemas CRISPR, en los que la interferencia está mediada por una única proteína grande (en este caso, Cas9) junto con las guías de ARN. De manera independiente, Jennifer Doudna y Emmanuelle Charpentier, recientes ganadoras del Premio Nobel de Química 2020, habían estado explorando a las proteínas asociadas a CRISPR para aprender cómo las bacterias utilizan a los espaciadores en sus sistemas inmunes. Juntas estudiaron un sistema CRISPR más simple que se basa en la proteína llamada Cas9. Encontraron que las bacterias responden ante un bacteriófago al transcribir espaciadores y ADN palindrómico en una larga molécula de ARN y que entonces la célula utilizaba un ARN llamado Trans-activating crRNA (tracrRNA) y también Cas9 para cortarla en pedazos. Al ser Cas9 una endonucleasa especializada en cortar ADN, tiene capacidad dos sitios de corte activos (HNH y RuvC), uno para cada cadena de la doble hélice. Parte de la investigación de las científicas recientemente galardonadas, fue que demostraron que podrían desactivar uno o ambos sitios preservando la habilidad de Cas9 de ser específico para su ADN objetivo. El equipo de investigación combinó al tracrRNA y ARN espaciador para formar una molécula llamada single-guide RNA que, al combinarse con la proteína Cas9, podía encontrar y cortar los blancos correctos de ADN (Figura 3). Con este conocimiento, propusieron que estos ARN guías podían sintetizarse para ser utilizados podrían utilizarse para la editar cualquier región en cualquier genoma, siempre y cuando ya se tuviera información del mismo. La primera vez que se demostró el sistema CRISPR-Cas9 funcionaba como una herramienta de ingeniería y edición genética en cultivos de células humanas, fue en el año 2012. Desde entonces, se ha utilizado en muchos organismos, incluidos los organismos modelo como la levadura del pan (Saccharomyces cerevisiae), el pez cebra (Danio rerio), la mosca de la fruta (Drosophila melanogaster), en el gusano Caenorhabditis elegans y en ratón (Mus musculus), así como en varias plantas, donde se ha conseguido su mejoramiento por medio de esta herramienta.
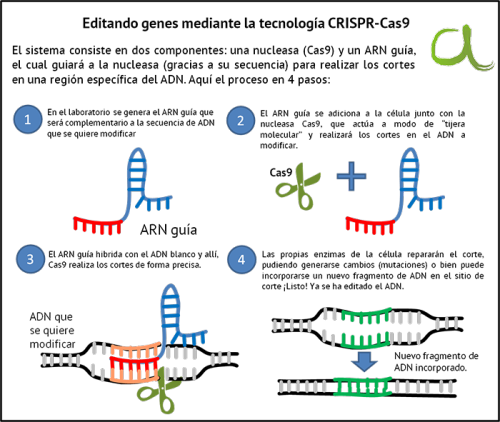
Figura 3. Esquema del mecanismo del sistema CRISPR.-Cas9 que funcionan como un “copiar y pegar” de la información genética.
El futuro y la controversia de la edición genética en humanos
La tecnología CRISPR-Cas9 ha tenido un impacto revolucionario en el mundo de la medicina. Según señalan en la Academia Sueca de Ciencias que otorga el premio Nobel, la edición genética solía ser un trabajo lento, difícil y, a veces, imposible. Sin embargo, el uso de la técnica CRISPR-Cas9 permite cambiar ‘el código de la vida’ en tan solo unas semanas. La primera evidencia de que CRISPR puede revertir síntomas de enfermedad en organismos vivos fue demostrada en marzo de 2014, cuando investigadores del Massachusetts Institute of Technology, curaron a ratones de desórdenes genéticos del hígado. Pero la gran esperanza es que puede permitir nuevas terapias para los seres humanos (Figura 4). Ya se han emprendido ensayos de tratamientos de la anemia de células falciformes, el cáncer o la ceguera hereditaria que se basan en CRISPR. Sin embargo, como se publicó en una entrega pasada (http://www.acmor.org.mx/?q=content/la-necesidad-de-congruencia-entre-la-ciencia-y-la-%C3%A9tica-la-edici%C3%B3n-gen%C3%A9tica), la edición genética en humanos es sin duda, un tema de debate debido a las implicaciones que esto puede tener.
Figura 4. Edición de células germinales en humanos con fines de corregir errores que produzcan enfermedades genéticas.
Desde la perspectiva científica, la edición de genomas aún tiene limitantes. Aunque tenemos la capacidad de corregir enfermedades hereditarias con los cual se solucionarían grandes problemas de salud y sobre todo mejorar la calidad de vida de toda la humanidad, aún existen muchos riesgos desconocidos donde la generación de otros defectos o enfermedades genéticas que también serían heredables. Por lo tanto, regular las situaciones que ameritarían este tipo de terapia de ingeniería genética es de suma importancia. La edición de genomas en humanos, es aún un trabajo en proceso ya que, hasta ahora, se ha aprobado el uso de la tecnología CRISPR-Cas9 para la edición de embriones humanos, solo como último recurso para corregir errores genéticos que pudieran provocar enfermedades hereditarias. Entonces, no solo debemos mejorar o estar seguros de que la tecnología de edición de genomas funcione, sino también de que la relación entre la enfermedad y las variantes en el genoma estén fuertemente asociadas. Pero lo más difícil es garantizar que los cambios por la edición genética, no desencadenen otro problema y predecir esto, sigue siendo un reto para la ciencia.
Finalmente, el premio ha sido bien merecido por las científicas ganadoras, que aunque se han visto en litigios relacionados con la propiedad intelectual de la tecnología CRISPR-Cas9, su contribución científica a la humanidad, es indiscutible. Con respeco a la ética de la edición genética en humanos, existe un esfuerzo mundial con el cual se ha iniciado una organización no gubernamental (ONG) llamada Association for Responsible Research and Innovation in Genome Editing (ARRIGE; http://arrige.org/) y como su nombre en inglés nos sugiere, estaría encargada de promover la investigación responsable e innovar en el área de edición de genomas. El lanzamiento de esta organización se llevó a cabo en Paris, Francia en marzo del 2018 y desde entonces trata de informar a todos los involucrados (académicos, compañías privadas, ciudadanos y gente encargada de tomar decisiones) acerca del desarrollo de las tecnologías para edición de genomas para llevarla a cabo en un ambiente social de aceptación. Mientras esta tecnología no esté regulada o completamente disponible para cualquier persona, puede ser otro detonador de desigualdad social, lo cual sería una pena para la ciencia y la tecnología.
Esta columna se prepara y edita semana con semana, en conjunto con investigadores morelenses convencidos del valor del conocimiento científico para el desarrollo social y económico de Morelos. Desde la Academia de Ciencias de Morelos externamos nuestra preocupación por el vacío que genera la extinción de la Secretaría de Innovación, Ciencia y Tecnología dentro del ecosistema de innovación estatal que se debilita sin la participación del Gobierno del Estado.
Referencias
https://pubs.acs.org/doi/full/10.1021/jacs.8b01551?utm_source=pubs_content_marketing&utm_medium=axial&utm_description=PUBS_1020_JHS_2020chemnobel&ref=pubs_content_marketing_axial_PUBS_1020_JHS_2020chemnobel
https://www.broadinstitute.org/files/news/pdfs/PIIS0092867415017055.pdf
Ligas de interés
https://www.investigacionyciencia.es/noticias/premio-nobel-de-qumica-para-el-mtodo-crispr-cas9-de-edicin-del-genoma-19099
https://www.nobelprize.org/prizes/chemistry/2020/press-release/
https://www.broadinstitute.org/what-broad/areas-focus/project-spotlight/crispr-timeline
https://es.wikipedia.org/wiki/CRISPR










 Desarrollado por Linabat
Desarrollado por Linabat